Copernic et origines
Je reviens sur l'ADN mitochondrial. Les mitochondries sont de petites entités (on dit des « organites ») présentes dans chacune de nos cellules (hommes et femmes) et lieux de synthèses importantes. Il se trouve que ces mitochondries ont un ADN, indépendant de celui présent dans le noyau. L’ADN mitochondrial est distinct de celui des chromosomes. Le pourquoi d’une telle situation sort du cadre de cet article mais cet ADN est transmis d’une génération à l’autre, et ça c’est notre sujet. La taille de cet ADN est de 16569 nucléotides, ce qui est tout petit par rapport à la taille du plus petit de nos chromosome, le chromosome Y : 53 millions de nucléotides.
La transmission mère-fille ne doit surtout pas faire oublier que les hommes aussi ont des mitochondries et, donc, de l’ADN mitochondrial. Il se trouve que le spermatozoïde perd son flagelle en pénétrant dans l’ovule, et avec le flagelle ses mitochondries. Ce sont donc les mitochondries présentes dans l’ovule qui sont transmises aux enfants.
Ces rappels étant faits, on peut ajouter que la relative petite taille de l’ADN permet un séquençage total, la détermination exacte de l’enchainement des nucléotides avec des implications médicales car il y a des mutations de cet ADN qui sont responsables de maladies génétiques, et aussi des implications génétiques car, en se transmettant de mère en fille sans remaniement (les « crossing-over » qui affectent les chromosomes) de rares mutations (un nucléotide modifié en un autre, ou disparu ou dédoublé) se sont accumulées et permettent de retracer l’arbre des filiations. Une société commerciale spécialisée dans les tests génétiques a permis à de nombreux particuliers de connaître leur ADN mitochondrial et, par là, où ils se situent dans l’arbre génétique.
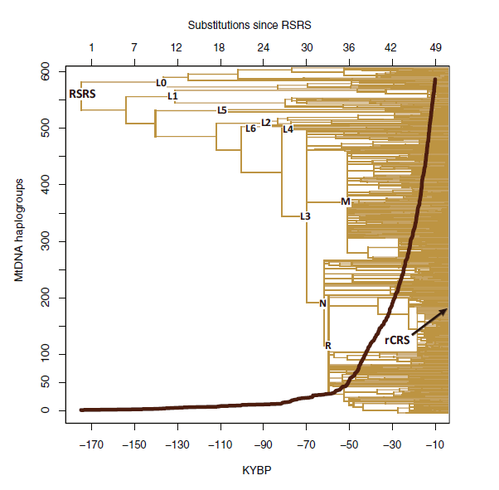
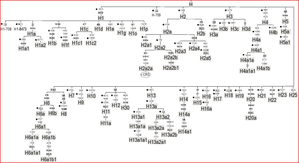
C’est justement l’ensemble de ces analyses « payantes », collectées (uniquement dans le cas où les personnes l’acceptent - bien sûr) dans une banque de données qui viennent de permettre une méga mise à jour en doublant (quasiment) le nombre de séquences, ce qui abouti à un arbre très détaillé et, un nouveau regard sur cet ADN qui justifie la référence à Copernic dans le titre de l’article publié (et de ce texte) : A Copernican reassessment of the human mitochondrial genome. Que vient faire Copernic dans cette galère ? Pour dire qu’une séquence comporte une mutation il faut la comparer, il faut une référence et, au tout début de cette quête c’est la séquence d’un quidam anglais à qui avait échu ce rôle. Il s’est vite avéré qu’il s’agissait d’une séquence très éloignée de la racine de l’arbre génétique tel qu’on pouvait le reconstruire. Dans l’article, le pas est franchi et sur la base notamment des séquences d’ADN mitochondriaux d’hommes de Neandertal ayant une position externe nécessaire pour « raciner » un arbre, une séquence théorique, RSRS, pour « Reconstructed Sapiens Reference Sequence » remplit maintenant ce rôle.
Sur la figure ci-dessus (extraite de l’article) la position de l’ancienne référence est rCRS (pour « revised Cambridge Reference Sequence »). Une observation très utile faite à partir des résultats (mais faite par d’autres avant cet article) a été que les substitutions, un type de mutations, en excluant par exemple les délétions et les duplications s’accumulent de façon quasi linéaire avec le temps, au rythme de 7 substitutions tous les 20000 ans ; c’est ce que les abscisses (en haut et en bas de la figure) représentent. Cette figure combine les résultats de 18843 ADN séquencés (KYBP = Kilo Years Before Present).
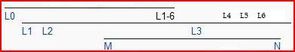
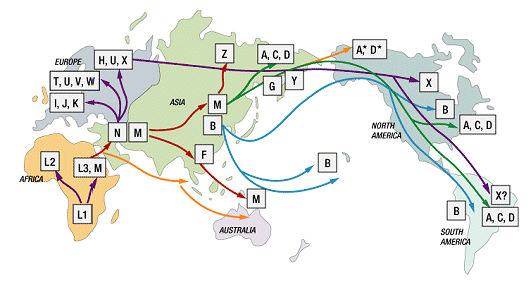
Par rapport à la théorie « Out of Africa » qui veut que la sortie d’Afrique se soit fait en une fois, c’est L3 qui est à la base de tous les ADNs hors d’Afrique avec 2 branches : M et N ; la branche M correspond à une migration « sud » longeant les côtes vers l’Inde puis l’Océanie et, vers le nord (sans doute) le passage vers les 2 Amériques , alors que la branche N aboutit aux groupes (on dit « haplogroupes ») couvrant l’Europe et le moyen orient. Le début de la « croissance exponentielle » figurée par le nombre de groupes, précède le néolithique et est plutôt contemporain de la conquête de l’Europe (et d’autres territoires nordiques) à partir de -50000 ans. Tous les autres groupes L, L0 à L6 sont spécifiques à l’Afrique, sauf pour ce qui suit sur le groupe L1b. L0 est un groupe qui est typique des populations ayant une langue à clic (voir le film : « Les dieux sont tombés sur la tête »).
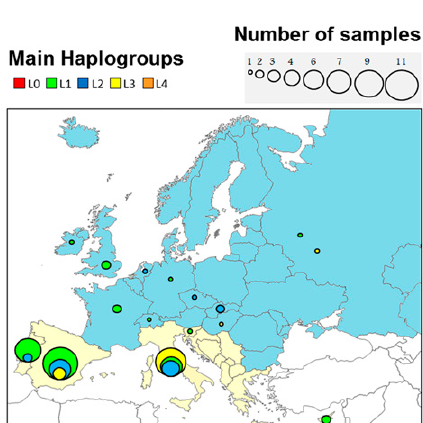
Il se trouve que mon attention a été attirée par un autre article portant sur la présence à très basse fréquence d’haplogroupes L en Europe. Bien entendu, il y a eu toute une série de mouvements de populations assez récents, dont la traite des esclaves, qui expliquent pour partie ces résultats inattendus. En filtrant ces cas, on trouve quelques cas, une cinquantaine à partir de la base de données non encore augmentée (9400 ADN) formant des groupes avec des mutations spécifiques (non connues en Afrique) et dispersés à travers l’Europe.
Extrait de : Cerezo et al. (2012) ; 10.1101/gr.134452.111
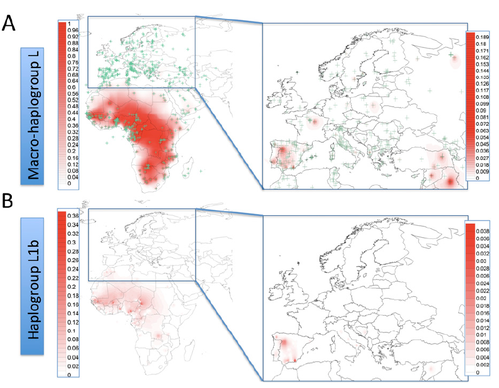
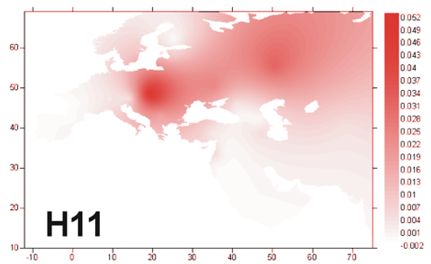
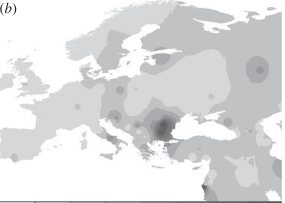
La figure ci-dessus montre la répartition de l’ensemble des haplogroupes L en Afrique puis du sous groupe L1b, l’une des deux branches de L1 avec L1c . Il s’agit de groupes dont l’origine est très ancienne et, même les « rameaux » comme L1b1a sont anciens. Par rapport, toujours, à « Out of Africa » la question posée est celle de mouvements de populations, anciens, et à l’écart des 2 branches M et N qui, selon ce modèle, sont les seuls à diffuser dans le reste du monde à partir de l’Afrique. La carte ci-dessous est un peu plus précise :
Ce qui est frappant, c’est la dispersion de l’haplogroupe L1b (et quelques L2) à 0.5 % ou même moins, à travers l’Europe. Ce que suggèrent ces données c’est une ancienne arrivée de L1b1a en Europe et évolution sur place. Le groupe L1b1a8, par exemple, est inconnu sur le continent africain alors qu’il y a déjà 6 cas distincts connus ayant de telles différences que cela indique un ancêtre commun il y a 10000 ans (pour ce seul groupe). Si l’arrivée de ces L1b1a correspond à une seule migration elle doit être bien antérieure.
Ce que cet article veut montrer c’est que le nombre de séquences mitochondriales connues est maintenant suffisant pour permettre des observations fines. Avec le doublement de la base de données, l’article de Cerezo et al. aurait plus d’une centaine de cas à discuter. Behar et al. (auteurs de l’article « A Copernican… ») trouve que l’haplogroupe U, issu de R (voir la figure de l’arbre génétique) est apparu il y a 45000 ans ce qui en fait un candidat sérieux mais j’ai voulu montrer que la génétique des populations nous réserve encore des surprises et une ancienne migration L1b1a expliquant l’une des cultures paléolithiques reste possible, égratignant au passage « Out of Africa ». Il est quand même stupéfiant que des personnes de très lointaine origine européenne se découvrent d’un haplogroupe, jusque là considéré comme typique de l’Afrique équatoriale ; nous sommes tous cousins !
Pour ceux qui sont tentés : voir http://www.familytreedna.com ; ils ne sont pas les seuls mais ils sont leaders pour le séquençage .


















 On peut comprendre cette expansion qui va bien au delà de la zone Bantou proprement dite en considérant que les forgerons dans bien des ethnies ont été des Bantous, une sorte de caste de forgerons.
On peut comprendre cette expansion qui va bien au delà de la zone Bantou proprement dite en considérant que les forgerons dans bien des ethnies ont été des Bantous, une sorte de caste de forgerons.